那就拿这篇最近的,便宜点的Eur Rev Med Pharmacol Sci(IF:3.784)分的文章来给你们讲讲,靶基因要怎么找转录因子……
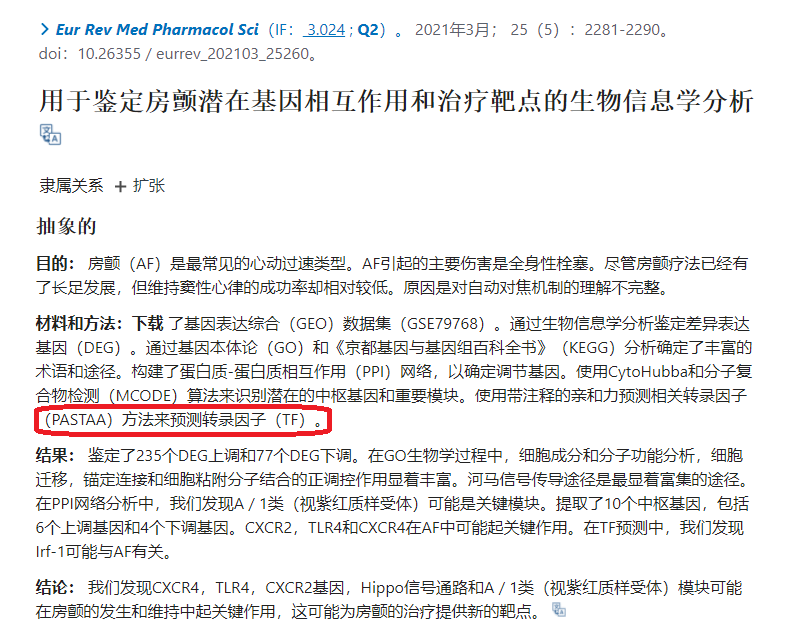
这篇文章主要内容就是生信分析,其中对于转录因子的预测用了一种叫PASTAA的算法……为啥是算法呢?其实没有一个工具叫PASTAA,我查了一下发在Bioinformatics(IF:6.931)上的文章,差不多就是一种算法:

差不多就是一种共表达和启动子结合比对的交集……
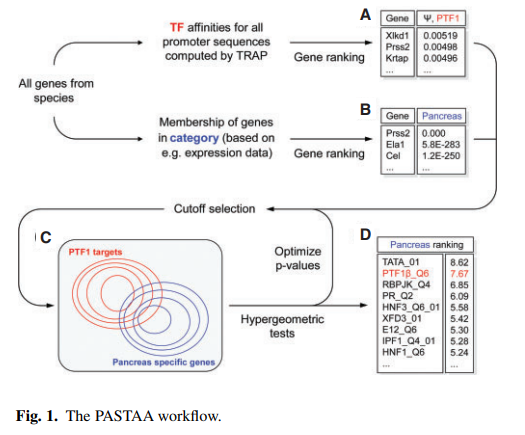
于是我翻了一下PASTAA算法中重要的工具……TRAP:

这个是用来测试转录因子和启动子间亲和力的,随便输入一个启动子序列:
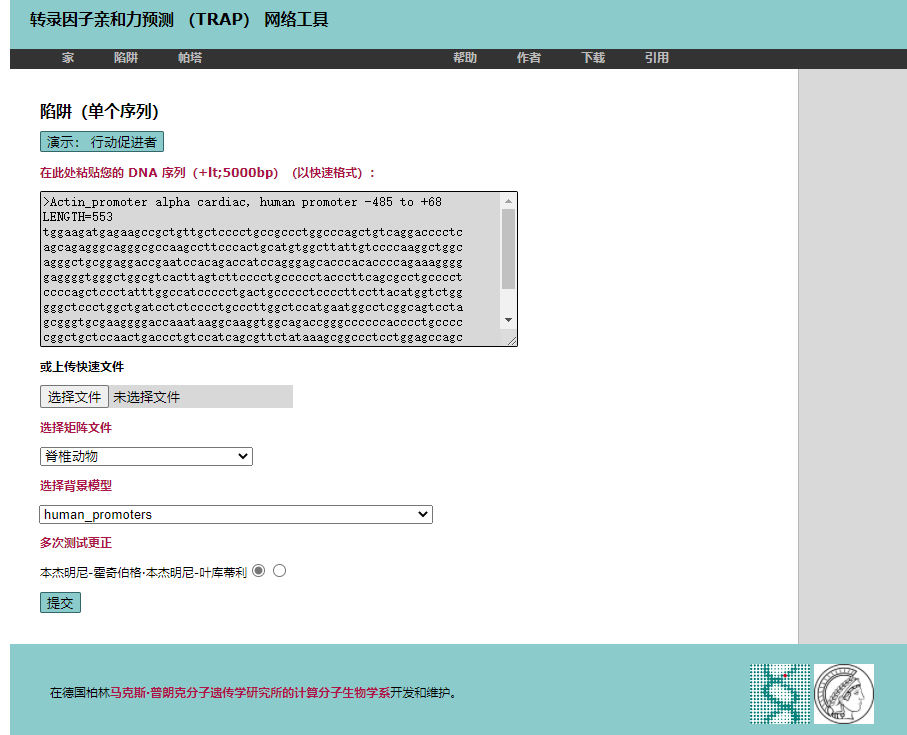
就可以预测对应启动子了,出来的是列表:
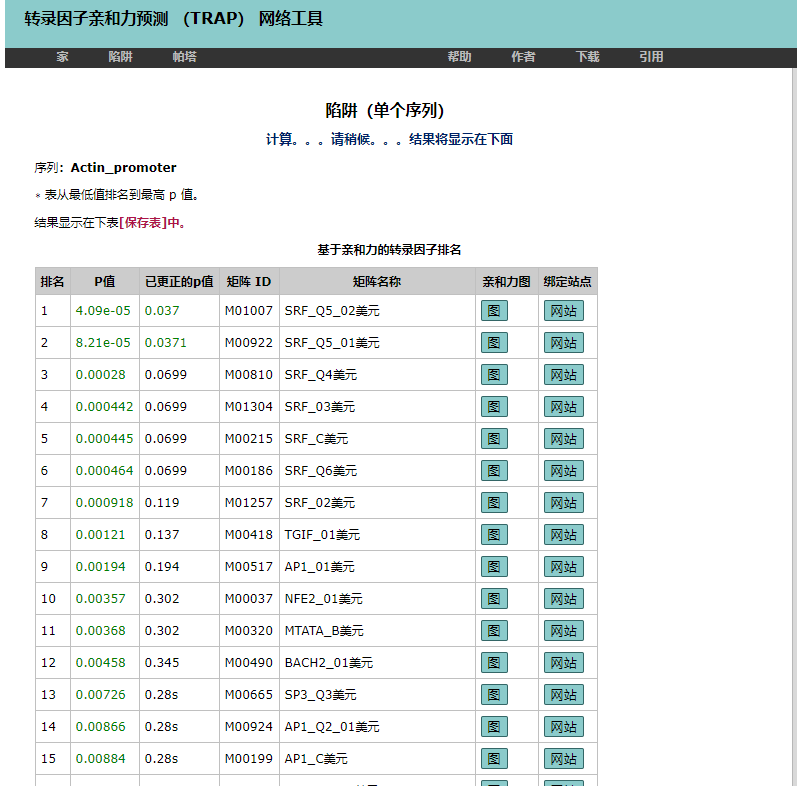
单击列表上的转录因子,就能显示具体的结合位置了:
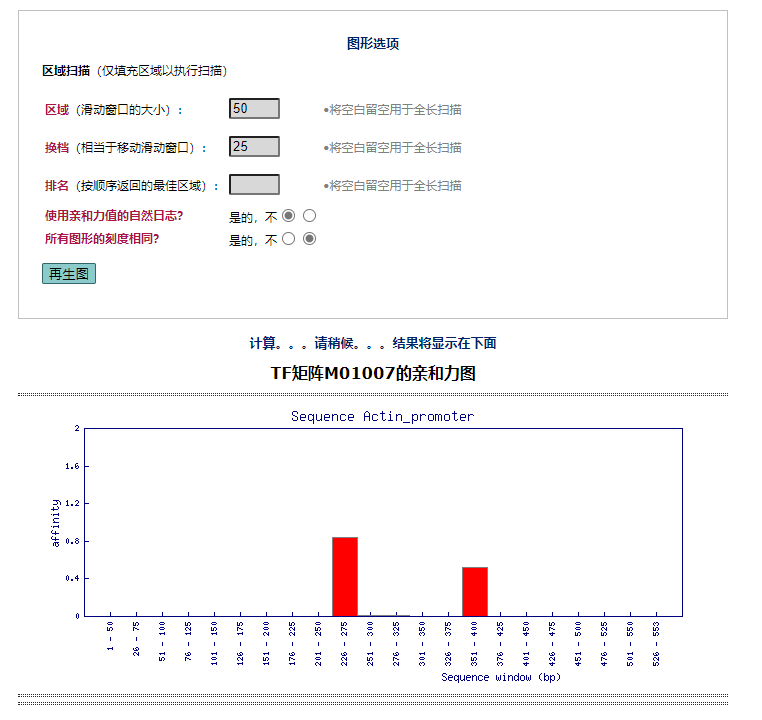
以及结合的具体序列……

第二个功能叫意大利面……是用来分析一系列基因的对应转录因子的,就只需要输入基因名就行了:
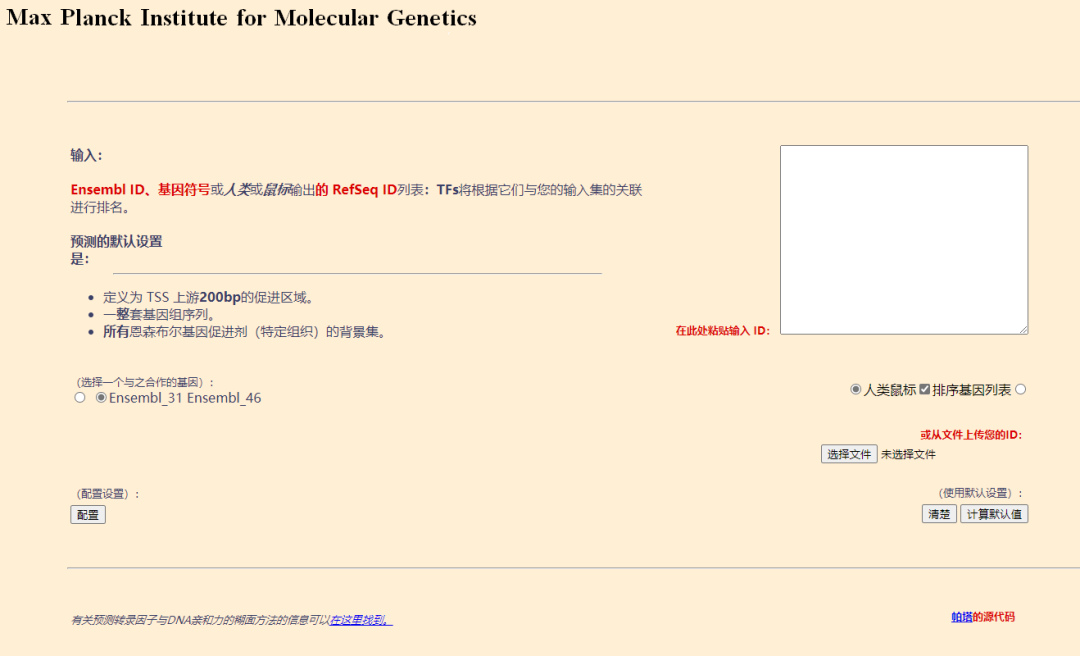
结果是你输入基因所对应的转录因子列表:
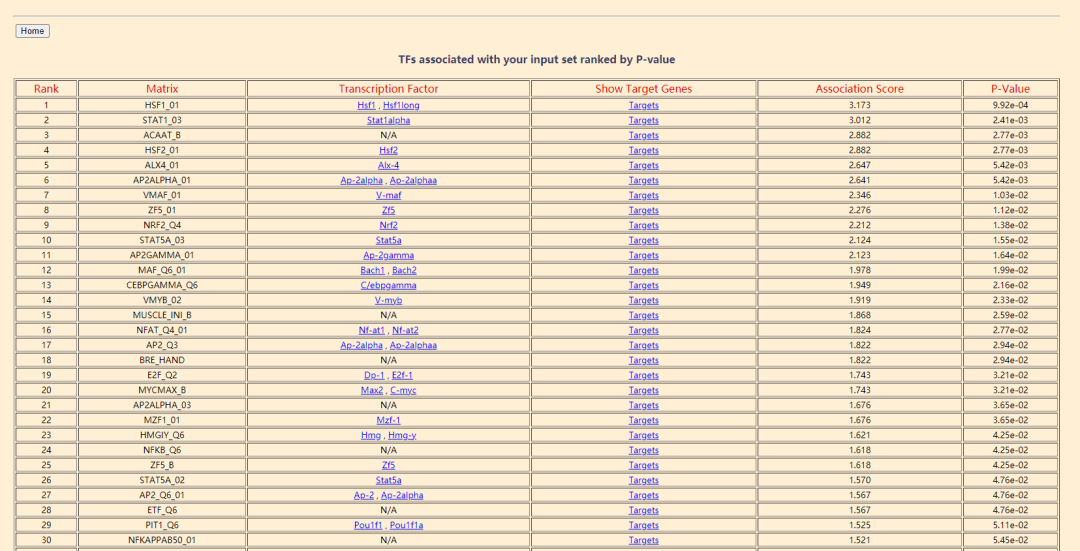
随便打开一个,显示的就是三个基因对应的转录因子的交集……
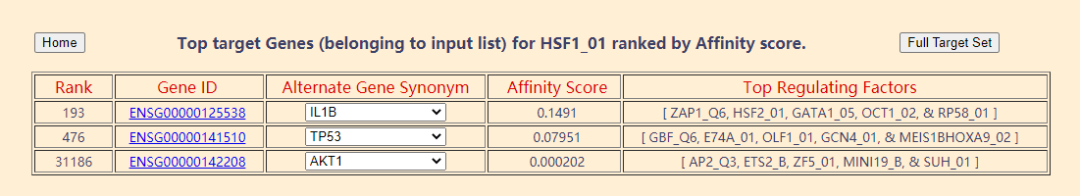
最有意思的一个功能叫SNP和转录因子……
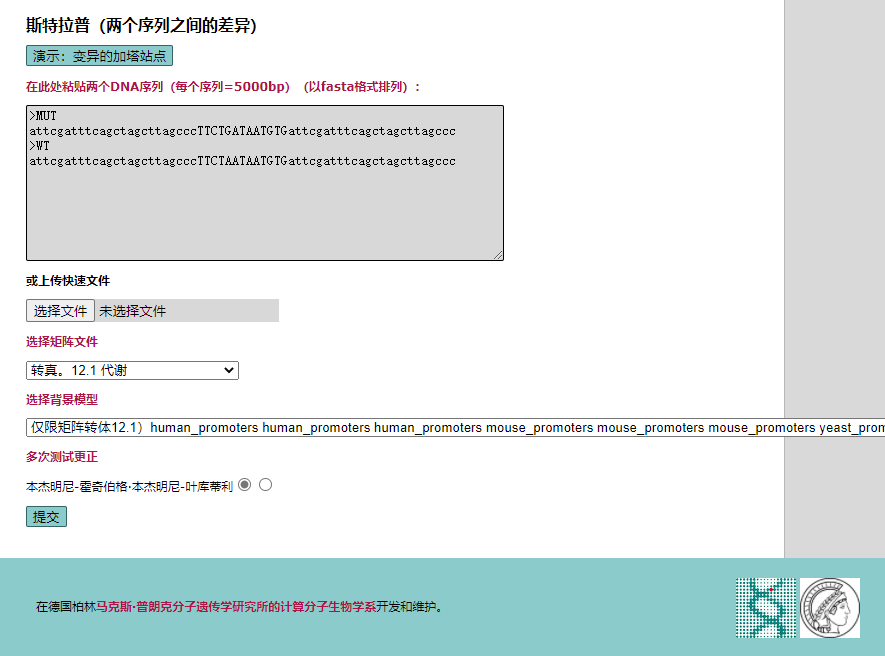
也就是分析单个碱基变化和转录因子亲和力之间的差异,输入相对应的野生型和突变型序列后,就能分析出两者结合上去转录因子能力的差异了: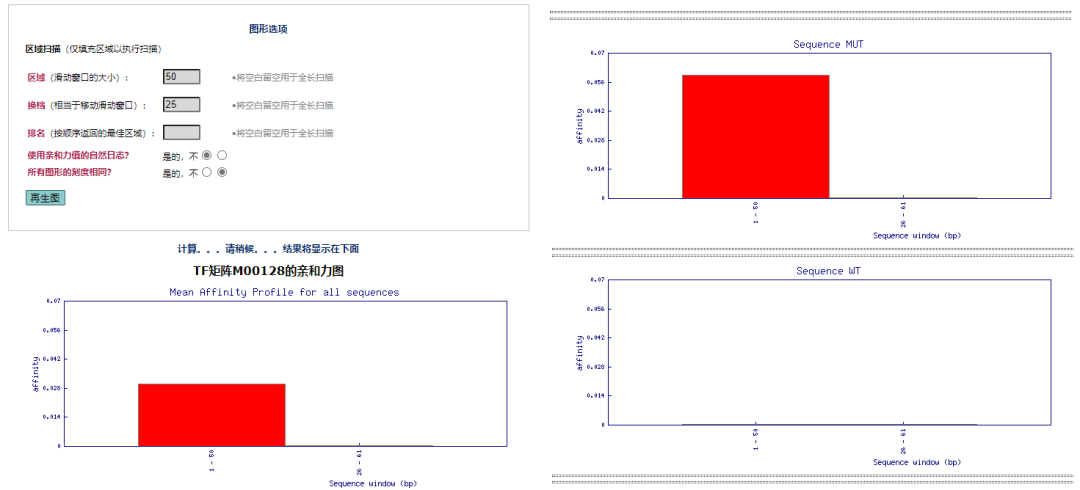
Bioinformatics | 有一个基因,要研究上游的转录因子要怎么办?
版权声明:
(1) 本文仅代表原作者观点,不代表本网站立场,请批判性阅读!
(2) 本文内容若存在版权问题,请联系我们及时处理。
(3) 除特别说明,原创内容版权归食用菌精准育种实验室所有,欢迎转载,转载请注明出处!
食用菌精准育种实验室 · Laboratory of Mushroom Precision Breeding