蛋白质磷酸化是生物学中最广泛的翻译后修饰之一【1,2】,其中丝氨酸、苏氨酸、酪氨酸和组氨酸残基上的蛋白质磷酸化几乎控制着真核细胞各个功能,迄今,基于质谱的磷酸化蛋白质组学技术已帮助人们鉴定出90000个丝氨酸(Ser)和苏氨酸(Thr)磷酸化位点,其中数千个与人类疾病和生物过程有关。解读这些蛋白激酶信号的必须手段就是阐明其下游的效应底物,然而到目前,对于绝大多数的磷酸化事件,人们都不知道这300多种的Ser/Thr激酶参与了哪些信号转导通路?它们的底物究竟是谁?这大大限制了人们对细胞磷酸化网络的理解,也限制了药物靶点的开发。
众所周知,激酶识别基序可以促进人们对新底物的发现,早年Lewis C Cantley和Benjamin E Turk发表的组合肽库筛选方法能够基于肽底物的磷酸化快速确定单个激酶的特异性【3,4】。
近日,来自美国威尔康奈尔医学院的Lewis C Cantley、麻省理工学院Michael B. Yaffe、和耶鲁大学Benjamin E Turk研究小组再度合作,在Nature杂志上发表题为An atlas of substrate specificities for the human serine/threonine kinome的研究论文,这篇文章在前期方法的基础上确定Ser/Thr激酶的最佳特异性底物,基于基序表征激酶之间的关系,构建了人细胞中300多种蛋白激酶的综合图谱。并使用全基因组数据集来计算注释和鉴定能够磷酸化人Ser/Thr磷酸化蛋白质组中每个报道的磷酸化位点的激酶,结果显示作者所预测的和报道的非常吻合。这项研究揭示了人Ser/Thr激酶组的内在底物特异性,为细胞信号转导过程中的磷酸化事件及生物学过程的研究带来了更多的资源。

现如今癌症基因组测序数据浩如烟海,而这其中却寻觅不到蛋白激酶与信号通路的相关研究,假如这些信息被人类所发现,对于疾病的治疗将会带来更多线索与契机。作者首先通过位置扫描肽阵列分析(PSPA),确定了人Ser/Thr激酶的底物识别基序,成功获得了303个Ser/Thr激酶的磷酸化位点基序(图1)。
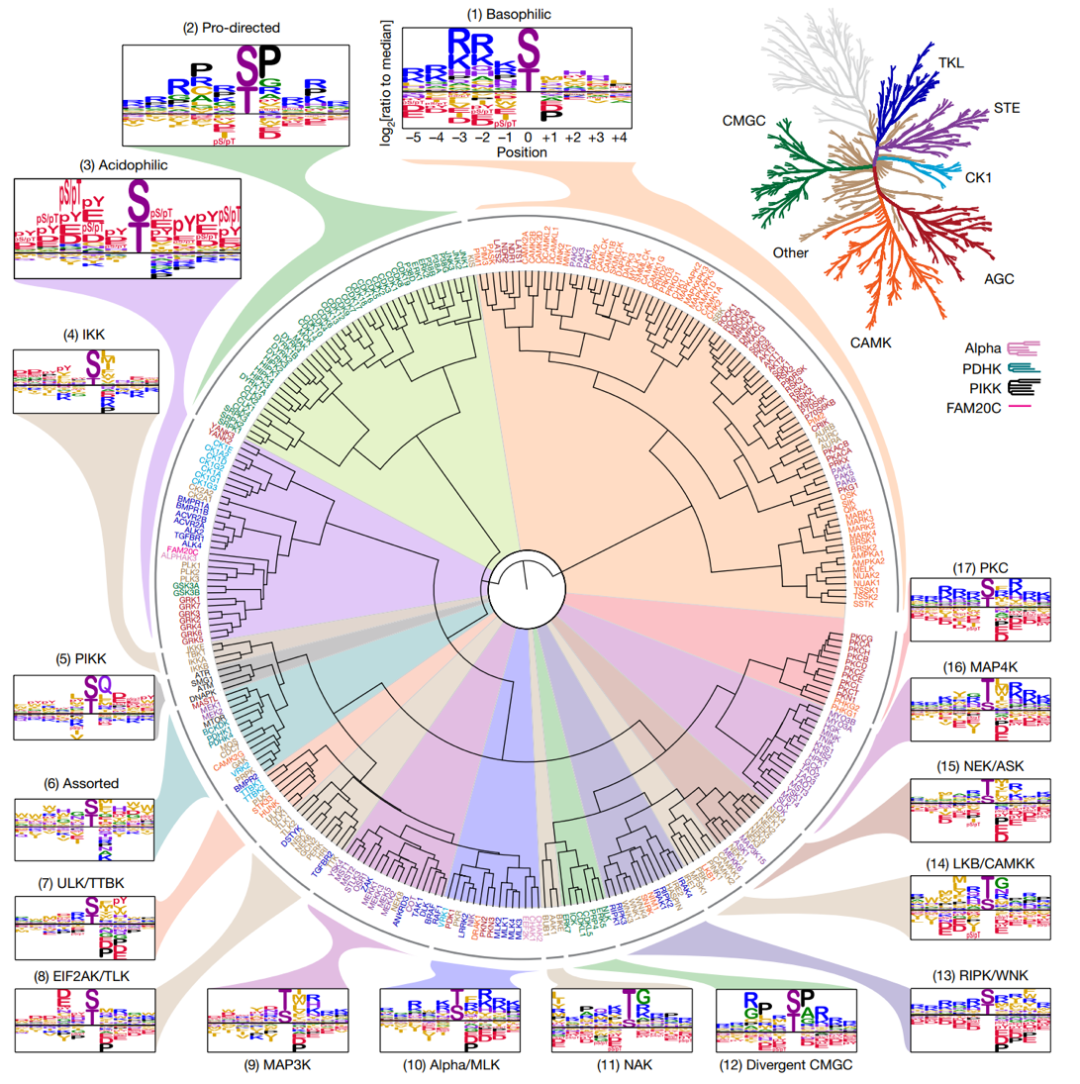 图1:人丝氨酸/苏氨酸激酶组的磷酸化位点基序树。
图1:人丝氨酸/苏氨酸激酶组的磷酸化位点基序树。
作者对磷酸化蛋白质组学结果进行全基因组注释,发现大多数情况下,磷酸化位点的线性序列背景对激酶-底物关系发挥重要的贡献。
值得注意的是,该基序预测还成功识别了磷酸化激酶研究中的首个里程碑事件【5】——1959年,Edwin G.Krebs和Edmond H.Fischer两位诺奖得主发现的糖原磷酸化酶的Ser15磷酸化,以及迄今为止报道最多的ATM-p53 Ser15磷酸化互作。
由于细胞信号转导网络的复杂性和动态性,作者需要对磷酸化蛋白质组进行全局基序分析,作者使用PLK1抑制剂处理HeLa细胞,含有PLK1基序的底物显著下调,另外ATM和ATR磷酸化的底物上调,与先前报道的结果高度一致,PLK1能够抑制有丝分裂细胞中的DNA损伤信号【6.7】。
另外,作者还分析了复杂干预条件所导致的信号变化事件,例如在电离辐射情况下,参与DNA损伤反应的经典激酶如ATM、ATR、DNA-PK上调,而参与细胞周期进展的经典激酶如CDK1、CDK2、CDK4、CDK6下调,与G1/S和G2/M阻滞一致。
此外,时间尺度的磷酸化变化也能够被该方法所解析,例如胰岛素刺激1 min后,MAPK通路激活,AMP激活的蛋白激酶在60分钟内下调。
总体而言,在这篇文章中,作者使用合成肽文库来分析了303 种Ser/Thr激酶的底物序列特异性,这种新的激酶图谱能够有助于研究人员识别正常和疾病状态下不同的信号通路,通过激酶来解码信号网络变化,应用这些信息能够捕获遗传学、药理学、代谢和环境变化后细胞和组织中信号通路的复杂改变,即便在疾病驱动基因不明确的情况下,也有可能帮助识别出驱动疾病发生的信号通路。
原文链接:
https://doi.org/10.1038/s41586-022-05575-3
参考文献
1. Cohen, P. The origins of protein phosphorylation. Nat. Cell Biol. 4, E127–E130 (2002).
2. Manning, G., Whyte, D. B., Martinez, R., Hunter, T. & Sudarsanam, S. The protein kinase complement of the human genome. Science 298, 1912–1934 (2002).
3. Songyang, Z. et al. Use of an oriented peptide library to determine the optimal substrates of protein kinases. Curr. Biol. 4, 973–982 (1994).
4. Hutti, J. E. et al. A rapid method for determining protein kinase phosphorylation specificity. Nat. Methods 1, 27–29 (2004).
5. Fischer, E. H., Graves, D. J., Crittenden, E. R. S. & Krebs, E. G. Structure of the site phosphorylated in the phosphorylase b to a reaction. J. Biol. Chem. 234, 1698–1704 (1959).
6. van Vugt, M. A. et al. A mitotic phosphorylation feedback network connects Cdk1, Plk1, 53BP1, and Chk2 to inactivate the G2/M DNA damage checkpoint. PLoS Biol. 8, e1000287 (2010).
7. Macůrek, L. et al. Polo-like kinase-1 is activated by aurora A to promote checkpoint recovery. Nature 455, 119–123 (2008).